Microbial Community Structure and Function and Their Influencing Factors in the Soil of Horqin Area of Tongliao City, Inner Mongolia
-
摘要: 农业作为通辽科尔沁地区的传统产业,是当地重要经济产业之一.了解土壤微生物群落结构和功能及其影响因素对农作物种植、污染土地修复等具有重要意义.采用Illumina Miseq高通量测序技术、FAPROTAX.1.1功能预测平台等方法,在该区采集71个土壤样品,分析其地球化学特征和微生物群落特征并对微生物群落进行功能预测,同时探讨不同环境因子和空间因子对微生物群落的影响.结果显示:微生物群落由变形菌门(Proteobacteria)(19.77%)、奇古菌门(Thaumarchaeota)(17.85%)、酸杆菌门(Acidobacteria)(17.14%)和放线菌门(Actinobacteria)(15.58%)等构成;功能预测表明该区存在大量活跃的参与氮循环过程的微生物功能群,其中有氧氨氧化功能群为该区的优势微生物功能群.方差分解分析显示,在对微生物群落结构差异的可解释范围内环境因子比空间因子的解释量更大.Mantel检验结果显示,整体微生物群落与土壤pH、EC、TN、C/N及Mg、Na、Sr元素含量显著相关(p < 0.05).综合分析表明,pH、EC和Sr元素含量是影响通辽科尔沁地区土壤微生物群落结构和功能及多样性的主要环境因素.Abstract: As a traditional industry in the Horqin area of Tongliao City,agriculture plays a pivotal role in the development of economy. Understanding the structure and function of soil microbial communities and their influencing factors is of great significance for crop planting and remediation of contaminated land. In this study,microbial community and function composition were investigated in a total of 71 soil samples collected from the Horqin area of Tongliao City with an integrated approach including 16S rRNA gene-based Illumina Miseq high-throughput sequencing technology and FAPROTAX.1.1 functional prediction platform,followed by statistical analysis with the use of R software package. The results show that the microbial communities in the studied soils were mainly composed of Proteobacteria (19.77%),Thaumarchaeota (17.85%),Acidobacteria (17.14%),Actinobacteria (15.58%). The functional prediction indicates that a large portion of the microbial communities in the studied soils were involved in nitrogen cycling,among which aerobic ammonia oxidation function predominated. Variation partition analysis (VPA) indicates that environmental factors accounted much more for the shaping of microbial community structure in interpretable range than spatial factors. Mantel test shows that the microbial communities in the studied soils were significantly (p < 0.05) correlated with soil pH,electrical conductivity (EC),total nitrogen (TN),ratio of carbon to nitrogen,and concentration of Mg,Na and Sr elements. It is concluded that pH,EC and Sr element concentrations were important environmental factors affecting the microbial community composition,function and diversity in the soils of the Horqin area of Tongliao City.
-
Key words:
- soil /
- microbial community /
- population composition /
- function /
- environmental response /
- ecology
-
0. 引言
土壤微生物是土壤中元素循环、有机质分解、土壤矿化等地球化学循环过程中的重要参与者(Guan et al., 2013),也是土壤物质循环的主要驱动者,参与并维持农业土壤的健康和生产力(Kennedy and Smith, 1995).土壤微生物的多样性对土壤生态系统的功能、可持续性和完整性至关重要,近年来越来越多的研究表明,较高的微生物多样性可以增强生态系统和微生物功能的稳定性(Garbeva et al., 2006;Chaer et al., 2009).不同功能微生物在不同类型作物生长、肥料利用、抑制病虫害等过程中都发挥着重要作用,如不同类型植物轮作会使土壤中微生物群落结构表现出明显的差异(Zhao et al., 2014);施用不同浓度的抗生素会使水稻田中的微生物群落结构产生不同的变化(Uddin et al., 2019);种植不同植物可改善重金属污染的土壤环境,提高土壤中微生物的活性(Wang et al., 2017;亓琳等,2018).因此,土壤微生物群落构成是衡量土壤健康的重要指标之一(Chu et al., 2007).健康优良的土壤环境是农作物生长的重要基础,了解土壤微生物群落结构和多样性及其影响因素对农作物种植、污染土地修复等具有重要意义.
生态环境变化会直接或间接地影响微生物群落结构和多样性,进而对整个生态系统产生影响.能够显著影响微生物群落结构变化的环境因素有土壤的质地类型(Tang et al., 2012)、含水率(刘洋等,2016)、电导率(赵萌等,2018)、pH(Shen et al., 2013)、元素含量(Khan et al., 2010)及植被类型(Li et al., 2018)等.此外,近年来有学者发现地理隔离也是影响土壤微生物群落分布的重要因素(Stopnisek et al., 2014;马转转等,2018).然而,环境因素和地理空间因素对微生物群落组成和多样性的影响贡献大小很难一概而论,但二者对微生物群落的影响都不容忽视.
农业作为通辽地区的主要支柱产业之一,在当地经济发展中发挥着举足轻重的作用.通辽地区科尔沁左翼中旗新开河流域是重要的粮食和蔬菜生产基地,食品安全与人民健康息息相关.在2016年开展的“内蒙古通辽地区多目标区域地球化学调查”中,中国地质大学(武汉)环境学院在新开河流域发现了近20 km2足硒-富硒土地,这是内蒙古东部地区首次发现的富硒土地.本研究重点分析该区农田土壤微生物群落及其与土壤元素等环境条件的关系,以期为合理开发利用土地、调整农业种植结构提供科学依据.本文采用高通量16S rRNA测序方法研究该地区土壤微生物群落构成特征,采用FAPROTAX.1.1功能预测平台预测了主要微生物功能特征,采用统计分析方法揭示了环境因子和空间因子对土壤微生物群落和功能构成的影响,从而为该地区农业的健康持续发展提供土壤微生物方面的基础资料.
1. 材料与方法
1.1 样品采集
土壤样品采集于2017年10月.采样区为通辽科尔沁左翼中旗宝龙山镇、架玛吐镇、努日木镇(附图 1),位于N43.5 ~44.5 、E122.2 ~123.5 (附表 1).该区属于北温带半干旱大陆性气候,多年平均气温5.5 ℃,平均降雨量为396.3 mm,平均蒸发量1 755.1 mm.采样区面积为600 km2,土地类型大部分为田地,少部分为草地和林地.采集土壤表层0~15 cm处土样,采样过程中尽量去除石头和根系.将采集的土壤样品分成2份,一份放在冰袋中保存用于土壤理化性质分析;另一份用50 mL的无菌离心管收集置于干冰箱保存,用于后续的微生物分析,运回实验室后放在-80 ℃条件下保存.
1.2 理化指标的测定
在澳实分析检测(广州)有限公司,使用方法代码ME⁃MS61r、四酸消解、质谱/光谱仪综合定量测试土壤样品的元素指标(表 1).在中国地质大学(武汉)材化学院通过元素分析仪测试土壤中总碳(TC)、总氮(TN)、总硫(TS)和总氢(TH);用燃烧法通过Vario TOC总有机碳分析仪(Elementar, Germany)测试土壤样品总有机碳(TOC);用双通道多参数水质分析仪(HQ40D, Field Case cat. No:58258⁃00,HACH,USA)测试土壤pH和电导率.
表 1 采样区71个土壤样品理化指标Table Supplementary Table Geochemical parameters of 71 soil samples in the Tongliao area参数 最小值 最大值 平均值 pH 6.61 9.91 7.76 EC (μS/cm) 18.63 1083 145.48 C/N 13.07 165.83 41.11 C/H 2.1 6.39 3.68 TN (%) 0.01 0.13 0.06 TC (%) 0.62 6.03 2.42 TS (%) 0.02 0.27 0.05 TH (%) 0.25 1.38 0.64 TOC (%) 0.21 2.82 0.86 Ca (mg/g) 9.7 55.1 23.8 Fe (mg/g) 13.4 35.1 20 K (mg/g) 18.4 25.6 23 Mg (mg/g) 3.5 12.1 6.5 Na (mg/g) 11 23.6 16.8 P (μg/g) 200 1 740.00 476.34 Zn (μg/g) 30 117 48.48 Mn (μg/g) 362 807 486.69 Cu (μg/g) 6.4 28.1 13.42 Ge (μg/g) 0.02 0.82 0.37 Ni (μg/g) 9.2 35.8 18.06 Pb (μg/g) 15.3 24.1 18.22 Se (μg/g) 0.06 0.23 0.1 Sr (μg/g) 208 369 260.42 As (μg/g) 3 16.7 7.76 Cd (μg/g) 0.03 0.2 0.11 Cr (μg/g) 20 52 32.28 Ga (μg/g) 11.6 20.2 14.31 1.3 DNA提取、PCR扩增及高通量测序
使用Fast DNA SPIN Kit for Soil试剂盒(MP Biomedical, Solon, OH, USA)提取土壤样品DNA.使用带barcode标记的高通量测序通用引物515F(5’⁃GTGYCAGCMGC CGCGGTA⁃3’)和806R(5’⁃CCCCGYCAA TTCMTTTRAGT⁃3’)(Zhang et al., 2018)对16S rRNA基因进行PCR扩增.PCR扩增体系:10×buffer 2.5 μL,2 mol/L dNTP 2 μL,515F和806R各1 μL,rTaq 0.3 μL,DNA模板1 μL,BSA 0.3 μL,用ddH2O补足至25 μL.PCR扩增程序:预变性95 ℃、5 min;变性95 ℃、45 s,退火53 ℃、30 s,延伸72 ℃、90 s,变性、退火和延伸重复39个循环;末次延伸72 ℃、10 min,最后在12 ℃下保存.采用1.5%的琼脂糖凝胶电泳对PCR产物进行检测,切取目的条带后用试剂盒(Axygen, Union City, DE, USA)进行纯化回收.使用NanoDrop ND⁃1000分光光度计对回收后的PCR产物进行浓度检测,然后送至Illumina Miseq测序平台进行高通量测序.
1.4 数据分析
参照Lynch and Neufeld(2015)的方法对获得的高通量序列进行筛选和过滤.通过FLASH(fast length adjustment of short reads)的默认设置对扩增后的序列进行拼接,拼接后将前后引物删除,用QIIME v1.8.0进行序列解析和高质量序列的筛选.使用USEARCH程序对序列进行嵌合体检测.采用UPARSE⁃OTU算法以97%的序列相似性划分分类单元(OTUs,operational taxonomic units)(Edgar, 2013).每个OTU的代表性序列的分类通过与RDP(ribosomal data base project)分类数据库比对来划分.
1.5 统计学分析
采用FAPROTAX.1.1(http://www.zoology.ubc.ca/louca/FAPROTAX/lib/php/index.php)数据平台对整体微生物群落进行功能预测(Louca et al., 2016).本文的统计分析使用origin 9.1和R语言(3.4.4节)完成.其中,Pearson相关性分析用“ggplot2”和“Himsc”包,CCA、PCNM、VPA和Mantel分析均使用“vegan”包进行.
1.6 登录序列号
本研究所得序列已提交至NCBI的SRA(sequence read archive)数据库,序列号为PRJNA518029.
2. 结果
2.1 研究区土壤理化性质
采样区土壤的pH为6.61~9.91,总体偏碱性.采样区土壤中的Ca(9.70~55.10 mg/g)、Na(11.00~23.60 mg/g)、Sr(208.00~369.00 μg/g)、As(3.00~16.70 μg/g)、Cd(0.03~0.20 μg/g)和Ni(9.20~35.80 μg/g)元素平均含量略高于全国土壤背景值;Cu(6.40~28.10 μg/g)、Fe(13.40~ 35.10 mg/g)、P(200.00~1 740.00 μg/g)、Pb(15.30~24.10 μg/g)、Mn(362.00~807.00 μg/g)、Mg(3.50~12.10 mg/g)、Zn(30.00~117.00 μg/g)、Cr(20.00~52.00 μg/g)、Ga(11.60~20.20 μg/g)、Ge(0.02~0.82 μg/g)和Se(0.06~0.23 μg/g)元素含量略低于全国土壤背景值(魏复盛等,1991)(表 1).总体而言,采样区土壤中营养元素特征表现为“富钾、缺磷、缺氮”.其中,K元素在采样区土壤中丰富,仅在宝龙山镇北侧和架玛吐镇西侧相对较低;P元素在采样区的东南部严重缺乏;N元素在全区表现为缺乏和相对缺乏,但在努日木镇较高.主要常量元素中,Na元素含量在研究区内变化较小,Ca和Mg元素含量变化较大,表现为:Na元素含量在宝龙山镇地区相对较高;Ca元素含量在宝龙山镇北侧、架玛吐镇西侧较高,而在架玛吐镇东南侧较低;Mg元素含量在宝龙山镇北侧、架玛吐镇西侧和东侧、努日木镇北侧较高.对于部分稀有元素,研究区大部分属于缺硒土壤,硒元素含量相对较高区域主要分布在架玛吐镇附近以及宝龙山镇和架玛吐镇之间小部分区域.
2.2 研究区土壤微生物群落分布特征
对通辽地区采集的土壤样品的高通量数据进行分析、处理,去除序列数低于5 000的样品后得到71个高质量样品的序列信息,共1 142 620条序列(附表 2).这些序列分属于35个门、96个纲、202个目、297个科和480个属.微生物群落主要由变形菌门(Proteobacteria)(19.77%)、奇古菌门(Thaumarchaeota)(17.85%)、酸杆菌门(Acidobacteria)(17.14%)、放线菌门(Actinobacteria)(15.58%)、芽单包菌门(Gemmatimonadetes)(5.4%)和拟杆菌门(Bacteroidetes)(5.2%)等构成.在纲级和目级水平上,这些序列分属于Nitrososphaeria(17.85%)、Gammaproteobacteria(9.51%)、Alphaproteobacteria (7.88%)、Subgroup 6(7.65%)、Blastocatellia(Subgroup 4)(7.49%)、Actinobacteria(5.36%)等纲和Nitrososphaerales、Pyrinomonadales、Betaproteobacteriales、Marine Group II、Tepidisphaerales、Gemmatimonadales等目.
功能预测结果显示可预测到的功能微生物群共有51种(附表 3).参与功能预测的每条OTU序列,有的归属于一种功能,有的可归属于多种功能.平均相对丰度 > 1%的微生物功能主要包括有氧氨氧化、硝化、硝酸盐还原和化能异养.预测结果表明该区存在大量参与氮循环功能的微生物群,其中有氧氨氧化功能群是硝化菌群的主要组成部分,也是该区的优势微生物功能群(图 1).硝化作用通常分为两步进行,包括有氧氨氧化和亚硝酸盐氧化过程,且有氧氨氧化过程是主要限制因素.
2.3 土壤微生物群落构成和功能对环境因子和空间因子的响应
2.3.1 土壤微生物群落构成和功能对环境因子的响应
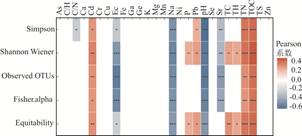
研究区各采样点土壤微生物群落的α多样性见附表 2.总体来看,采样区土壤微生物群落α多样性较高.其中,Shannon Wiener多样性指数为3.630~6.512,Simpson多样性指数为0.913~0.997,Equitability指数为0.908~0.632,Fisher指数为73.776~639.745,Observed OTUs为314~1407.多样性和环境因子的Pearson相关性分析结果显示,TOC、TN及Cd元素含量与α多样性呈显著性正相关,pH、EC及Na、Sr元素含量与α多样性呈显著性负相关(图 2).另外,微生物群落和单一环境因子的Mantel分析结果显示(附表 4),仅pH、EC、TN、C/N及Mg、Na、Sr元素含量与整体微生物群落表现出显著相关性(p < 0.05).
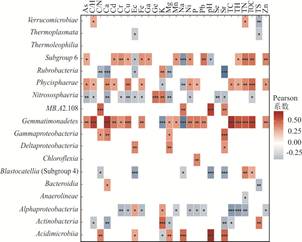
为进一步了解该区土壤微生物群落的主要门和纲分类单元对不同环境因子的响应,选取相对丰度 > 1%的微生物门和纲分类单元与环境因子进行Pearson相关性分析(图 3,图 4),结果显示同一环境因子对不同门、纲分类单元的影响有明显差异.例如,在Proteobacteria门中,Alphaproteobacteria纲与多种环境因子(TC、TN、TH及P、Cr、Cu、Fe、Mn、Ni、Pb、Zn元素含量)具有显著负相关关系,而Gammaproteobacteria纲和Deltaproteobacteria纲与Mg、Sr元素含量呈显著正相关,与其他环境因子无显著相关关系;Thaumarchaeota门及其门下的Nitrososphaeria纲与Ge、K元素含量呈显著正相关,与其他环境参数(C/H、EC、TOC及As、Ca、Cr、Cu、Fe、Mg、Ni、Se、Sr元素含量)呈显著负相关;Acidobacteria门与C/N、EC、pH及Na、Sr元素含量呈显著负相关,与TN、TOC及Cd、Pb元素含量呈显著正相关,Acidimicrobiia纲则与C/N、EC、pH及Mg、Na、Sr元素含量呈显著正相关,与K元素含量为负相关关系.
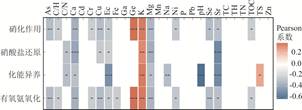
笔者对所预测的主要功能群的相对丰度和该区的环境因子进行Pearson相关性分析发现(图 5):有氧氨氧化菌群和硝化作用菌群与Ge、K元素含量呈显著正相关,与TOC、C/H、EC及Ca、Mg、As、Cr、Cu、Ni、Se、Sr元素含量呈显著负相关;化能异养菌群与TS、K元素含量呈显著正相关,与C/N、EC、pH及Ca、Mg、Na、Sr元素含量呈显著负相关;硝酸盐还原菌群与K元素含量呈显著正相关,与C/N、EC及Ca、Mg、Cu、Sr元素含量呈显著负相关.总体来看,EC及Ca、Mg、K、Sr元素含量分别与该地区的主要功能群显著相关.
2.3.2 土壤微生物群落构成和功能对空间因子的响应
通过PCNM分析可获得样品在空间关系上不同尺度的分解向量,与群落结构显著相关的PCNM因子即为群落在该尺度表现出的显著的空间结构(赵安玖等,2009).PCNM分析可得到PCNM1、PCNM2…PCNMn,通常认为PCNM1属于大尺度,代表整个研究尺度内空间因子的信息,PCNM2…PCNMn则描述越来越小的尺度信息(Borcard et al., 2004).PCNM分析在71个样点上产生了38个分解向量,筛选出与群落结构显著相关(p < 0.05)的PCNM因子为PCNM1和PCNM19;为了便于分析微生物群落在不同空间尺度的变化,在采样范围内划分PCNM1为大尺度、PCNM19为小尺度.空间因子与整体微生物群落及主要门、纲级别(相对丰度大于1%)的群落结构的Pearson相关性分析结果显示,小尺度(PCNM19)空间因子信息与整体微生物群落的相关性比大尺度(PCNM1)强,而与门、纲级别的微生物群落的相关性则相反(附表 5).
由附表 5可知,有氧氨氧化功能群(0.134 vs. 0.229)、硝化功能群(0.136 vs. 0.229)及硝酸盐还原功能群(0.181 vs. 0.240)与PCNM1的相关性均小于PCNM19,化能异养菌群与PCNM1(0.152)和PCNM19(0.152)的相关性无明显差异.表明小尺度空间因子(PCNM19)对功能群落的影响更大.其次,在整个空间内,随地理距离的增加微生物群落结构差异略微变大,但在小范围内群落分布差异较大(附图 2).
2.3.3 环境因子和空间因子对微生物群落构成和功能的相对重要性
将环境因子和空间因子去除自相关因子后(去除自相关之后的环境因子为pH、EC、Na、Sr和TN)与整体微生物群落进行典范对应分析(CCA)(图 6a),结果显示CCA1和CCA2的解释量分别为7.6%和4.0%,EC、Sr、Na、pH和TN以及空间因子(PCNM1和PCNM19)均为塑造微生物群落结构的重要影响因子.其次,采用VPA分析环境因子(去除自相关之后的环境因子)和空间因子(PCNM1和PCNM19)对整体微生物群落的相对贡献.图 6b显示环境因子和空间因子对群落结构贡献的总体解释量为17.94%、未解释量为82.06%;其中,环境因子的解释量占13.34%,空间因子的解释量占2.91%,两者的共同解释量占1.69%.总体来看,环境因子对整体微生物群落差异的贡献在可解释的范围内大于空间因子.
环境因子和空间因子对不同功能的微生物群落的贡献明显不同,有氧氨氧化菌群的VPA分析显示(图 7a),空间因子和环境因子对该群落结构变化总解释量为27.92%、未解释量为72.08%,其中环境因子和空间因子的单独解释量分别为22.42%和1.09%,两者共同的解释量为4.41%.环境因子和空间因子对硝化作用菌群(图 7b)的贡献和有氧氨氧化菌群差异很小,再次印证了有氧氨氧化作用为该区域硝化作用的主导过程.硝酸盐还原菌群中(图 7c),环境因子和空间因子对该功能群的解释量分别为15.73%和4.54%,两者共同的解释量占2.40%,未解释量占77.33%;化能异养菌群中环境因子和空间因子的解释量分别为12.87%和3.44%,两者共同的解释量占1.64%,未解释量占到了82.05%(图 7d).总体来看,仍有大量未知因素对功能群落的结构产生影响,但在本文数据可解释的范围内,环境因子对功能群的贡献大于空间因子.
3. 讨论
3.1 采样区土壤微生物的优势菌群
采样区土壤微生物的优势菌门为变形菌门,这与朱立月等(2018)对内蒙古地区土壤微生物群落优势细菌群的研究结果一致,但也有所不同:前人采用高通量测序的方法检测到土壤样品中Alphaproteobacteria、Betaproteobacteria、Deltaproteobacteria和Gammaproteobacteria的存在;而本文的样品中未检测到Betaproteobacteria,只检测到其余3种.Fierer et al.(2007)认为,Betaproteobacteria更倾向于在富营养的环境中生长,可能由于研究区土壤中氮磷元素普遍缺乏,导致Betaproteobacteria的生存处于劣势.前人在研究小麦-水稻和小麦-玉米轮作的实验中发现了类似现象,Betaproteobacteria在有效磷和总氮含量更高的水稻土壤中相对丰度要高于玉米土壤(Zhao et al., 2014).奇古菌门(Thaumarchaeota)是研究区土壤微生物的第二大优势菌门,其门下Nitrososphaeria纲为该区的优势菌纲.奇古菌门是最先从中温泉古菌门单独划分出的一个新的门类,后被发现广泛分布于海洋、森林、农田、湿地、沙漠土壤等环境中(Isobe et al., 2012;Marusenko et al., 2013;Xu et al., 2019),主要包括氨氧化古菌GroupⅠ.1a和Ⅰb、酸性土壤中的GroupⅠ.1c、开放海域的ALOHA Group、热泉和热液口的pSL12 Group和HWCGIII/Nitrosocaldus Group等(张丽梅和贺纪正,2012).本文的功能预测结果也显示研究区的奇古菌属于有氧氨氧化功能群和硝化功能群.总的来说,采样区土壤微生物群落组成的结果与前人在通辽其他地区的研究结果大致相同,但由于土地利用类型和植被及其外源的营养物质输入的不同,同一门类不同亚纲的相对丰度仍存在较大差异(Degrune et al., 2015;陈孟立等,2018)
3.2 采样区土壤微生物群落的影响因素
3.2.1 环境因素对土壤微生物群落的影响
环境因素是影响微生物群落特征的重要因素(Lauber et al., 2008; Brockett et al., 2012). CCA(图 6a)和Mantel(附表 4)结果证明在测得的环境变量中,土壤pH、EC和Sr元素含量对微生物群落的影响较大.前人在一些研究中也发现pH是影响微生物群落结构的重要因素(Lauber et al., 2009; Chu et al., 2010; Shen et al., 2013;唐阳等,2018;张艳敏等,2018),本文的结果显示土壤pH对该区总体微生物群落结构(OTU水平)和多样性影响显著(图 2,附表 4),其中Actinobacteria和Acidobacteria分别与pH表现出显著的正相关和负相关;前人在研究北美和南美地区88个不同生态类型的土壤样品微生物群落的主要门类的相对丰度时也发现Actinobacteria和Acidobacteria与pH有相似的关系(pH为4~9),变形菌纲对pH未表现出明显的相关性,这些都与本文的研究结果一致(图 3).另外,在其研究中还观察到Bacteroidetes与pH有明显的正相关关系(Lauber et al., 2009),而本研究中Bacteroidetes对pH并不敏感(图 3).虽然pH对研究区的总体微生物群落和多样性影响显著,但大部分门、纲水平的微生物群落的相对丰度对pH的响应并不明显(图 3,图 4).一方面,可能pH不是影响这些微生物相对丰度的主要因素,土壤盐度或碳氮等营养元素对微生物的限制作用更大;另一方面,土壤pH不是直接影响微生物的群落结构,而是作为一个综合指标,土壤的其他理化性质如有效氮磷的利用、土壤有机碳、金属离子的溶解性等均与pH有直接或间接的关系(Lauber et al., 2009).
通常用溶液电导率来间接测定和表征土壤的含盐量,故可用电导率间接指示土壤盐渍化程度(Pereira et al., 2019),前人研究认为土壤电导率是影响微生物群落结构的重要因素(赵萌等,2018;Uddin et al., 2019).本研究中EC和Na、Mg、Ca元素含量对微生物群落影响显著(图 2,附表 4),土壤电导率对微生物群落多样性和主要功能群都表现出潜在的抑制作用(图 2,图 5),表明研究区的微生物群落受到土壤盐渍化的影响.
Sr是碱土金属中丰度最低的元素,本研究中该元素含量对整体微生物群落的影响最为显著(附表 4),对α多样性和主要功能微生物群(图 2,图 5)表现出潜在的抑制作用,表明采样区微生物群落受到Sr污染的影响较严重.前人关于Sr污染对微生物群落影响的研究结果存在差异:亓琳等(2018)采用Biolog微平板方法研究燕麦修复Sr污染的土壤时发现,被Sr污染的土壤随Sr浓度的增高微生物群落多样性显著降低,种植燕麦可显著增加土壤中微生物群落功能多样性;但Wang et al.(2017)采用同样的方法对Sr污染的土壤的微生物进行培养检测,发现Sr(土壤有效Sr浓度为242.2 mg/kg)能够促进土壤微生物群落的多样性和活性,高粱对Sr元素有较好的吸收效果.这种差异可能是由于不同实验样品中土壤Sr的背景值不同,不同微生物群落耐Sr的能力也不同.本实验采用高通量16S rRNA的方法仅能检测土壤中细菌和古菌的群落,其次样品中还检测到很多不可培养的微生物类群,这与前人采用培养方法研究微生物群落对Sr的响应存在很大差异.Sr元素是营养元素还是污染元素取决于其在实际土壤中的可利用程度.
另外,研究区Cd、As元素含量也高于全国土壤平均值,Cd元素含量与整体群落的多样性及门、纲级别的微生物群落构成均表现出显著的正相关(图 2~4).Khan et al.(2010)研究认为Cd污染会显著抑制土壤中微生物活性并改变微生物的群落结构,Ding et al.(2017)研究认为重金属浓度较低时也可促进微生物的活性;本研究可能由于当地的Cd污染造成了部分区域土壤中Cd浓度较高,从而筛选出一些适应高Cd浓度的微生物群落,导致微生物α多样性增加.但由于高Cd污染的面积相对较小,适应高Cd的微生物群落的相对丰度较低,故在分析其对整体微生物群落的影响时,Cd并未表现出明显的相关性(附表 4).As元素含量在整个区域的分布较为均匀,对微生物群落的多样性难以产生较大差异,因此未表现出与整体微生物群落的显著相关性.而Se元素在研究区内普遍缺乏,仅在部分区域含量相对较高,故在整个区域内未发现Se与微生物群落之间明显的相关关系.
3.2.2 空间因素对土壤微生物群落的影响
近年来有学者研究发现空间因子也是影响微生物群落结构的重要因素(Martiny et al., 2006;Zhang et al., 2018).在本研究中,空间因素对具有氧氨氧化功能和硝化功能的微生物群落差异的解释量很低(图 7a、7b),在整个采样区内微生物群落结构随地理距离的变化不是很大(附图 2).前人对氨氧化古菌的研究显示大区域尺度下地理距离对群落的影响较大(Marusenko, 2013;Jiang et al., 2014),而研究区范围(0~600 km2)相对较小,故在区域内没有产生明显的地理隔离.空间因素对硝酸盐还原菌群和化能异样菌群的解释量略高于前两者(图 7c、7d),由于参与这两种功能的微生物类型较多,无法确切解释何种类型的微生物受空间的相对影响较大,需要进一步的分析证明.其次,总体微生物群落对空间因子的响应也低于环境因子(图 6b),尽管随地理距离的变化,微生物群落结构差异略微增加(附图 2),但总体上仍是环境因子对微生物群落的影响占主导地位.综上所述,一方面,当存在某种对微生物群落选择性较强的环境因子时,环境因子对微生物群落的影响会大于空间因子;另一方面,在本研究中有超过75%的群落结构变异性不能用环境变量和空间因子来解释,仍有更多未知的因素对该区的土壤微生物群落产生重要影响.
此外,本研究采用的高通量测序和功能预测方法也有一定的技术局限性,比如高通量的测序只能检测到属水平的群落,功能预测只能对纳入数据库的微生物功能群进行分析.因此,深入了解该区域微生物群落的具体功能还需进一步采取功能基因鉴定等方法进行研究.
4. 结论
(1)该区微生物多样性较高,共获得35个门、96个纲、202个目、297个科和480个属,主要为变形菌门(Proteobacteria)(19.77%)、奇古菌门(Thaumarchaeota)(17.85%)、酸杆菌门(Acidobacteria)(17.14%)、放线菌门(Actinobacteria)(15.58%)等.功能预测结果显示微生物群落以有氧氨氧化菌群、硝化菌群、硝酸盐还原菌群、化能异养菌群为主.
(2)环境因素对研究区土壤微生物群落和功能的影响大于空间距离.主要环境因素为土壤pH、EC和Sr元素,其中EC和Sr元素对微生物群落的多样性和丰度表现出明显的抑制作用,反映出该区微生物群落可能受土壤盐渍化和重金属污染的影响.
附表和附图见本刊官网(http://www.earth⁃science.net).
-
表 1 采样区71个土壤样品理化指标
Table 1. Geochemical parameters of 71 soil samples in the Tongliao area
参数 最小值 最大值 平均值 pH 6.61 9.91 7.76 EC (μS/cm) 18.63 1083 145.48 C/N 13.07 165.83 41.11 C/H 2.1 6.39 3.68 TN (%) 0.01 0.13 0.06 TC (%) 0.62 6.03 2.42 TS (%) 0.02 0.27 0.05 TH (%) 0.25 1.38 0.64 TOC (%) 0.21 2.82 0.86 Ca (mg/g) 9.7 55.1 23.8 Fe (mg/g) 13.4 35.1 20 K (mg/g) 18.4 25.6 23 Mg (mg/g) 3.5 12.1 6.5 Na (mg/g) 11 23.6 16.8 P (μg/g) 200 1 740.00 476.34 Zn (μg/g) 30 117 48.48 Mn (μg/g) 362 807 486.69 Cu (μg/g) 6.4 28.1 13.42 Ge (μg/g) 0.02 0.82 0.37 Ni (μg/g) 9.2 35.8 18.06 Pb (μg/g) 15.3 24.1 18.22 Se (μg/g) 0.06 0.23 0.1 Sr (μg/g) 208 369 260.42 As (μg/g) 3 16.7 7.76 Cd (μg/g) 0.03 0.2 0.11 Cr (μg/g) 20 52 32.28 Ga (μg/g) 11.6 20.2 14.31 -
Borcard D., Legendre P., Avois-Jacquet C., et al.2004. Dissecting the Spatial Structure of Ecological Data at Multiple Scales. Ecology, 85(7):1826-1832.https://doi.org/10.1890/03-3111 doi: 10.1890-03-3111/ Brockett B. F. T., Prescott C. E., Grayston S. J.. 2012. Soil Moisture is the Major Factor Influencing Microbial Community Structure and Enzyme Activities across Seven Biogeoclimatic Zones in Western Canada. Soil Biology and Biochemistry, 44(1): 9-20. https://doi.org/10.1016/j.soilbio.2011.09.003 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=d0532e75a979fc1cf813ed8009af109f Chaer G., Fernandes M., Myrold D., et al. 2009. Comparative Resistance and Resilience of Soil Microbial Communities and Enzyme Activities in Adjacent Native Forest and Agricultural Soils. Microbial Ecology, 58(2): 414-424. https://doi.org/10.1007/s00248-009-9508-x http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=87fdf61b563f4f8a0584733c4948f18d Chen M.L., Zeng Q.C., Huang Y.M., et al. 2018. Effects of the Farmland-to-Forest/Grassland Conversion Program on the Soil Bacterial Community in the Loess Hilly Region. Environmental Science, 39(4): 1824-1832 (in Chinese with English abstract). http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=hjkx201804044 Chu H. Y., Fierer N., Lauber C. L., et al. 2010. Soil Bacterial Diversity in the Arctic is not Fundamentally Different from that Found in other Biomes. Environmental Microbiology, 12(11): 2998-3006. https://doi.org/10.1111/j.1462-2920.2010.02277.x Chu H. Y., Lin X. G., Fujii T., et al. 2007. Soil Microbial Biomass, Dehydrogenase Activity, Bacterial Community Structure in Response to Long-Term Fertilizer Management.Soil Biology and Biochemistry, 39(11): 2971-2976. https://doi.org/10.1016/j.soilbio.2007.05.031 Degrune F., Dufrêne M., Colinet G., et al. 2015. A Novel Subphylum Method Discriminates Better the Impact of Crop Management on Soil Microbial Community. Agronomy for Sustainable Development, 35(3): 1157-1166. https://doi.org/10.1007/s13593-015-0291-4 Ding Z.L., Wu J.P., You A.Q., et al.2017.Effects of Heavy Metals on Soil Microbial Community Structure and Diversity in the Rice (Oryza Sativa L. subsp. Japonica, Food Crops Institute of Jiangsu Academy of Agricultural Sciences) Rhizosphere. Soil Science & Plant Nutrition, 63(1):75-83. https://doi.org/10.1080 /00380768.2016.1247385 Edgar R. C.. 2013. UPARSE: Highly Accurate OTU Sequences from Microbial Amplicon Reads. Nature Methods, 10(10): 996-998. https://doi.org/10.1038/nmeth.2604 http://d.old.wanfangdata.com.cn/NSTLQK/NSTL_QKJJ0231145773/ Fierer N., Bradford M. A., Jackson R. B.. 2007. Toward an Ecological Classification of Soil Bacteria. Ecology, 88(6): 1354-1364. https://doi.org/10.1890/05-1839 doi: 10.1890-05-1839/ Garbeva P., Postma J., van Veen J. A., et al. 2006. Effect of Above-Ground Plant Species on Soil Microbial Community Structure and Its Impact on Suppression of Rhizoctonia Solani AG3. Environmental Microbiology, 8(2): 233-246. https://doi.org/10.1111/j.1462-2920.2005.00888.x http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=4a758f5903563a1eab7cbe29e78d52bb Guan X. Y., Wang J. F., Zhao H., et al. 2013. Soil Bacterial Communities Shaped by Geochemical Factors and Land Use in a Less-Explored Area, Tibetan Plateau. BMC Genomics, 14(1): 820. https://doi.org/10.1186/1471-2164-14-820 Isobe K., Koba K., Suwa Y., et al. 2012. High Abundance of Ammonia-Oxidizing Archaea in Acidified Subtropical Forest Soils in Southern China after Long-Term N Deposition. FEMS Microbiology Eco-logy, 80(1): 193-203. https://doi.org/10.1111/j.1574-6941.2011.01294.x http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=7aca8b3c486924085ab2c033448b4459 Jiang H.C., Hang L.Q., Deng Y., et al.2014.Latitudinal Distribution of Ammonia-Oxidizing Bacteria and Archaea in the Agricultural Soils of Eastern China. Applied and Environmental Microbiology, 80(18):5593-5602.https://doi.org/10.1128/aem.01617-14 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=4c3c209e7e343f50cb08195de03794a0 Kennedy A. C., Smith K. L.. 1995. Soil Microbial Diversity and the Sustainability of Agricultural Soils. Plant and Soil, 170(1): 75-86. https://doi.org/10.1007/bf02183056 http://d.old.wanfangdata.com.cn/OAPaper/oai_doaj-articles_4241b49ddefcd4e7b3340e0fbe6d9562 Khan S., El-Latif Hesham A., Qiao M., et al. 2010. Effects of Cd and Pb on Soil Microbial Community Structure and Activities. Environmental Science and Pollution Research, 17(2): 288-296. https://doi.org/10.1007/s11356-009-0134-4 doi: 10.1016-j.ecoleng.2011.07.002/ Lauber C.L., Hamady M., Knight R., et al.2009.Pyrosequencing-Based Assessment of Soil pH as a Predictor of Soil Bacterial Community Structure at the Continental Scale. Applied Environmental Microbiology, 75(15):5111-5120.https://doi.org/10.1128/aem.00335-09 doi: 10.1128-AEM.00335-09/ Lauber C. L., Strickland M. S., Bradford M. A., et al. 2008. The Influence of Soil Properties on the Structure of Bacterial and Fungal Communities across Land-Use Types. Soil Biology and Biochemistry, 40(9): 2407-2415. https://doi.org/10.1016/j.soilbio.2008.05.021 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=59bd9fb69b2ee0a0f7d22a2f6e0d75d6 Li N., Shao T. Y., Zhu T. S., et al. 2018. Vegetation Succession Influences Soil Carbon Sequestration in Coastal Alkali-Saline Soils in Southeast China. Scientific Reports, 8(1):9728-9739.https://doi.org/10.1038/s41598-018-28054-0 Liu Y., Huang Y.M., Zeng Q.C.. 2016. Soil Bacterial Communities under Different Vegetation Types in the Loess Plateau. Environmental Science, 37(10): 3931-3938 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/Periodical/hjkx201610035 Louca S., Parfrey L. W., Doebeli M.. 2016. Decoupling Function and Taxonomy in the Global Ocean Microbiome. Science, 353(6305): 1272-1277. https://doi.org/10.1126/science.aaf4507 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=2f05c6bb2e6feefcc778ff870b43476a Lynch M. D. J., Neufeld J. D.. 2015. Ecology and Exploration of the Rare Biosphere. Nature Reviews Microbiology, 13(4): 217-229. https://doi.org/10.1038/nrmicro3400 Ma Z.Z., Qiao S.S., Cao M.W., et al. 2018.Environmental Selection and Dispersal Limitation Drive the Assemblage of Bacterial Community in Temperate Forest Soils. Chinese Journal of Applied Ecology, 29(4): 1179-1189 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/Periodical/yystxb201804019 Martiny J. B., Bohannan B.J., Brown J.H., et al. 2006.Microbial Biogeography: Putting Microorganisms on the Map. Nature Reviews. Microbiology, 4(2): 102-112. https://doi.org/10.1038/nrmicro1341 http://d.old.wanfangdata.com.cn/OAPaper/oai_doaj-articles_bf3d74a0fb5c4b90c8e46eb4b560fc0a Marusenko Y., Bates S.T., Anderson I., et al. 2013. Ammonia-Oxidizing Archaea and Bacteria are Structured by Geography in Biological Soil Crusts across North American Arid Lands. Ecological Processes, 2(1):9-19. https://doi.org/10.1186/2192-1709-2-9 Pereira C.S., Lopes I., Abrantes I., et al.2019.Salinization Effects on Coastal Ecosystems: A Terrestrial Model Ecosystem Approach. Philosophical Transactions of the Royal Society B:Biological Sciences, 374(1764):20180251. https://doi.org/10.1098/rstb.2018.0251 Qi L., Li Y.L., Zhao W., et al. 2018. Effect of Avena Sativa L. on Soil Enzyme Activity and Microbe Functional Diversity under Strontium Pollution. Acta Ecologica Sinica, 38(13): 4888-4896 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/OAPaper/oai_doaj-articles_eff7ec6f64ead86411810ab6de60daa2 Shen C. C., Xiong J. B., Zhang H. Y., et al. 2013. Soil pH Drives the Spatial Distribution of Bacterial Communities along Elevation on Changbai Mountain. Soil Biology and Biochemistry, 57: 204-211. https://doi.org/10.1016/j.soilbio.2012.07.013 Stopnisek N., Bodenhausen N., Frey B., et al.2014.Genus-wide Acid Tolerance Accounts for the Biogeographical Distribution of Soil Burkholderia Populations. Environmental Microbiology, 16(6):1503-1512.https://doi.org/10.1111/1462-2920.12211 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=3320fa16cc5b60c79d328e3e3ae7b1e3 Tang J., Ding X., Wang L.M., et al. 2012. Effects of Wetland Degradation on Bacterial Community in the Zoige Wetland of Qinghai-Tibetan Plateau (China). World Journal of Microbiology and Biotechnology, 28(2): 649-657. https://doi.org/10.1007/s11274-011-0858-4 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=d008553d9eb8ae19f365295109aed9d6 Tang Y., Liu Y.C., Yang J., et al. 2018. Gene Diversity Involved in Kalvin Pathway of Carbon Fixation and Its Response to Environmental Variables in Surface Sediments of the Northern Qinghai-Tibetan Plateau Lakes. Earth Science, 43(S1): 19-30 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/Periodical/dqkx2018z1003 Uddin M., Chen J. W., Qiao X. L., et al. 2019.Bacterial Community Variations in Paddy Soils Induced by Application of Veterinary Antibiotics in Plant-Soil Systems. Ecotoxicology and Environmental Safety, 167: 44-53. https://doi.org/10.1016/j.ecoenv.2018.09.101 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=ffab20446ac91d45e4f1d1079c8a3aca Wang X., Chen C., Wang J.L.. 2017. Phytoremediation of Strontium Contaminated Soil by Sorghum Bi-Color (L.) Moench and Soil Microbial Community-Level Physiological Profiles (CLPPs).Environmental Science and Pollution Research, 24(8): 7668-7678. https://doi.org/10.1007/s11356-017-8432-8 Wei F.S., Yang G.Z., Jiang D.Z., et al. 1991. Basic Statistics and Characteristics of Soil Element Background Value in China. Environmental Monitoring in China, 7(1): 1-6 (in Chinese). Xu Y., Wang H.M., Xiang X., et al.2019.Vertical Variation of Nitrogen Fixers and Ammonia Oxidizers along a Sediment Profile in the Dajiuhu Peatland, Central China.Journal of Earth Science, 30(2):397-406. https://doi.org/10.1007/s12583-018-0982-2 http://d.old.wanfangdata.com.cn/Periodical/dqkx-e201902015 Zhang L.M., He J.Z.. 2012. A Novel Archaeal Phylum: Thaumarchaeota: A Review. Acta Microbiologica Sinica, 52(4): 411-421 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/OAPaper/oai_pubmedcentral.nih.gov_2292581 Zhang Y.M., Wu G., Jiang H.C.. 2018. Research Progress on Microorganisms Driving Carbon Cycle in Hot Spring. Earth Science, 43(S1): 31-41 (in Chinese with English abstract). https://www.sciencedirect.com/science/article/pii/S1006810414600362 Zhang Y. M., Wu G., Jiang H. C., et al. 2018. Abundant and Rare Microbial Biospheres Respond Differently to Environmental and Spatial Factors in Tibetan Hot Springs. Frontiers in Microbiology, 9:2096. https://doi.org/10.3389/fmicb.2018.02096 Zhao A.J., Hu T.X., Chen X.H.. 2009. Multiple-Scale Spatial Analysis of Community Structure in a Mountainous Mixed Evergreen-Deciduous Broad-Leaved Forest, Southwest China. Biodiversity Science, 17(1): 43-50 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/OAPaper/oai_doaj-articles_8f2e64c39236cf52f1ca1283f30c029e Zhao J., Zhang R. F., Xue C., et al. 2014. Pyrosequencing Reveals Contrasting Soil Bacterial Diversity and Community Structure of Two Main Winter Wheat Cropping Systems in China. Microbial Ecology, 67(2): 443-453. https://doi.org/10.1007/s00248-013-0322-0 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=36526d24a86bfe57f22dc8b0730abe6b Zhao M., Yin C.S., Li C.W., et al. 2018. Using Miseq Sequencing to Analyze Seasonal Soil Microbial Community Dynamics in Reclaimed Scirpus Mariqueter Coastal Wetlands. Journal of Shanghai Ocean University, 27(5): 718-727 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/Periodical/shscdxxb201805009 Zhu L.Y., Ding M.Y., Huang G.N., et al. 2018. Microbial Community Structure and the Correlation between Environmental Factors and Community Structure in Sandy Soil of Tongliao in Inner Mongolia. Science Technology and Engineering, 18(20): 347-352 (in Chinese with English abstract). http://d.old.wanfangdata.com.cn/Periodical/kxjsygc201820052 陈孟立, 曾全超, 黄懿梅, 等. 2018.黄土丘陵区退耕还林还草对土壤细菌群落结构的影响.环境科学, 39(4): 1824-1832. http://d.old.wanfangdata.com.cn/Periodical/hjkx201804044 刘洋, 黄懿梅, 曾全超. 2016.黄土高原不同植被类型下土壤细菌群落特征研究.环境科学, 37(10): 3931-3938. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=hjkx201610035 马转转, 乔沙沙, 曹苗文, 等. 2018.环境选择和扩散限制驱动温带森林土壤细菌群落的构建.应用生态学报, 29(4): 1179-1189. http://d.old.wanfangdata.com.cn/Periodical/yystxb201804019 亓琳, 李艳玲, 赵威, 等. 2018.锶污染下燕麦对土壤酶活性和微生物群落功能多样性的影响.生态学报, 38(13): 4888-4896. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=stxb201813034 唐阳, 刘永超, 杨渐, 等. 2018.青藏高原北部湖泊表层沉积物参与卡尔文循环的固碳基因多样性及其影响因素.地球科学, 43(S1): 19-30. http://d.old.wanfangdata.com.cn/Periodical/dqkx2018z1003 魏复盛, 杨国治, 蒋德珍, 等. 1991.中国土壤元素背景值基本统计量及其特征.中国环境监测, 7(1): 1-6. http://www.cnki.com.cn/Article/CJFDTotal-IAOB199101000.htm 张丽梅, 贺纪正. 2012.一个新的古菌类群:奇古菌门(Thaumarchaeota).微生物学报, 52(4): 411-421. http://www.cnki.com.cn/Article/CJFDTotal-WSXB201204003.htm 张艳敏, 吴耿, 蒋宏忱. 2018.热泉中驱动碳循环的微生物研究进展.地球科学, 43(S1): 31-41. http://d.old.wanfangdata.com.cn/Periodical/dqkx2018z1004 赵安玖, 胡庭兴, 陈小红. 2009.西南山地阔叶混交林群落空间结构的多尺度特征.生物多样性, 17(1): 43-50. http://d.old.wanfangdata.com.cn/Periodical/swdyx200901005 赵萌, 印春生, 厉成伟, 等. 2018. Miseq测序分析围垦后海三棱藨草湿地土壤微生物群落多样性的季节变化.上海海洋大学学报, 27(5): 718-727. http://d.old.wanfangdata.com.cn/Periodical/shscdxxb201805009 朱立月, 丁美月, 黄冠南, 等. 2018.内蒙古通辽市砂质土壤中微生物群落结构及其与环境因子的关系.科学技术与工程, 18(20): 347-352. http://d.old.wanfangdata.com.cn/Periodical/kxjsygc201820052 期刊类型引用(9)
1. 王亚妮,胡宜刚,王增如,李以康,张振华,周华坤. 人工植被重建对沙化高寒草地土壤真菌群落特征的影响. 土壤学报. 2023(01): 280-291 .  百度学术
百度学术2. 朱子超,刘慧,毛胜军,马奥兰,李民敬. 河水-地下水侧向交互带微生物群落分布特征及其主控因子. 地球科学. 2023(10): 3832-3843 .  本站查看
本站查看3. 孙娇娇,朱卫红,金日,曹光兰. 图们江下游湿地不同植物群落区表层土壤中菌类的磷脂脂肪酸组成和含量. 湿地科学. 2023(05): 797-805 .  百度学术
百度学术4. 张攀,谢先军,黎清华,甘致远,胡甜,杨渐,邓娅敏,甘义群,张彦鹏. 东寨港红树林沉积物中微生物群落结构特征及其对环境的响应. 地球科学. 2022(03): 1122-1135 .  本站查看
本站查看5. 高明华,于春艳,刘金月,于凤莅,孙广霁. 呼伦贝尔草甸草原土壤古菌群落多样性高通量分析. 饲料博览. 2022(05): 8-15 .  百度学术
百度学术6. 张仲富,喻庆国,王行,刘会会,赵亚川,谢雪杨,张萌,耿玮. 植物群落和土壤理化性质对碧塔海湿地土壤细菌群落的影响. 应用生态学报. 2021(06): 2199-2208 .  百度学术
百度学术7. 陶彦臻,苏春利,谢先军,曾邯斌,潘洪捷,闫福贵. 基于碎石屏障的土壤盐渍化改良技术及机理研究. 地球科学. 2021(11): 4118-4126 .  本站查看
本站查看8. 刘俊,张世文,宋桂芳,王阳,祝亚飞,鲁胜军,兰淼,夏沙沙. 复垦土壤细菌群落结构及其与土壤肥力的关系. 农业工程学报. 2021(21): 124-133 .  百度学术
百度学术9. 李永恒,李乾玺,吴君君,程晓莉. 秦岭落叶阔叶林不同空间尺度下土壤微生物特征. 植物科学学报. 2020(03): 335-346 .  百度学术
百度学术其他类型引用(7)
-
 肖玉娜附表.docx
肖玉娜附表.docx

-









 下载:
下载:








 下载:
下载:



 百度学术
百度学术







